DNA kwantificeren
Hoeveel DNA zit er in een spoor?
Het antwoord op die vraag is lastig en vereist veel geduld en nauwkeurigheid
om deze vraag te beantwoorden. Een poging van het NFI (NFI-rapport dd. 21
april 2006) demonstreert dit overduidelijk:
Er kan een schatting worden gemaakt van het aantal cellen dat zich in
een bepaald biologisch spoor bevindt. In deze zaak zijn voorafgaand aan de
DNA-onderzoeken die zijn uitgevoerd in 2003 en 2004 de DNA-extracten van
de bemonsteringen van de blouse aan een semi-kwantitatieve DNA-test
onderworpen.(..) De semi-kwantitatieve test van het contactspoor van
bemonstering [ARA852]#20 van de blouse geeft aan dat de DNA-concentratie
in het extract van deze bemonstering ongeveer 0,03 nanogram (ng)
per microliter (Ál) bedraagt. De bemonstering [ARA852]#20 van de blouse is
indertijd in 400 Ál vloeistof geŰxtraheerd. Dit betekent dat de
bemonstering in totaal 400 x 0,03 = 12 ng DNA bevat. Bekend is dat de
hoeveelheid DNA in een lichaamscel ongeveer 5 picogram (pg; 1 ng is 1000
pg) bedraagt. Dus, 12 ng DNA komt uitgaande van een hoeveelheid DNA van 5
pg per cel neer op circa 2400 cellen (12 ng gedeeld door 5pg)."
Het gaat hier om een spoor, dat volgens de
NFI-gegevens met QIAamp werd geŰxtraheerd (meer hierover in paragraaf
6.1.1). De berekening werd echter gebaseerd op de veronderstelling, dat de
extractie met Chelex werd uitgevoerd. Bij gebruik van Chelex ontstaat 400
Ál extract, bij QIAmp echter 50-100 Ál, waardoor de berekening ineens 4
tot 8 x lager uitkomt. Ook is de hoeveelheid DNA in een cel eerder 7 dan 5
picogram.
Om meer zekerheid te verkrijgen omtrent de
hoeveelheid in een monster is naast de vergelijking van de uitkomsten van
de semi-kwanitatieve test ook een vergelijking gemaakt van de sterktes van
de profielen. De semi-kwantitatieve test kent slechts een beperkt aantal
afleesmogelijkheden (in stappen van x2), waardoor er eerder sprake is van
intervallen, dan van waarden. Eerst moet geadresseerd worden, in welke
mate de sterktes van profielen maatgevend zijn voor de hoeveelheid
aangetroffen DNA.
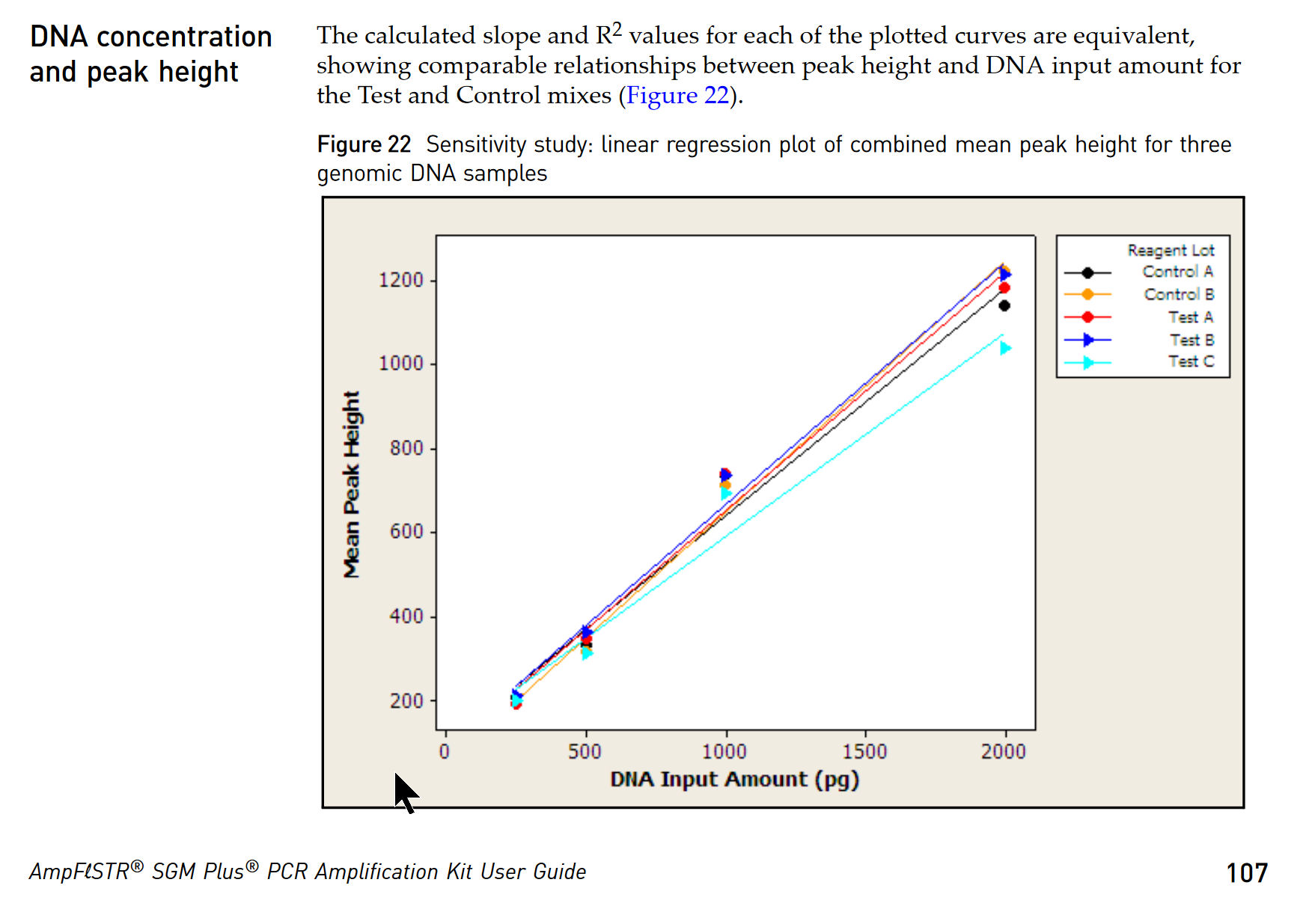
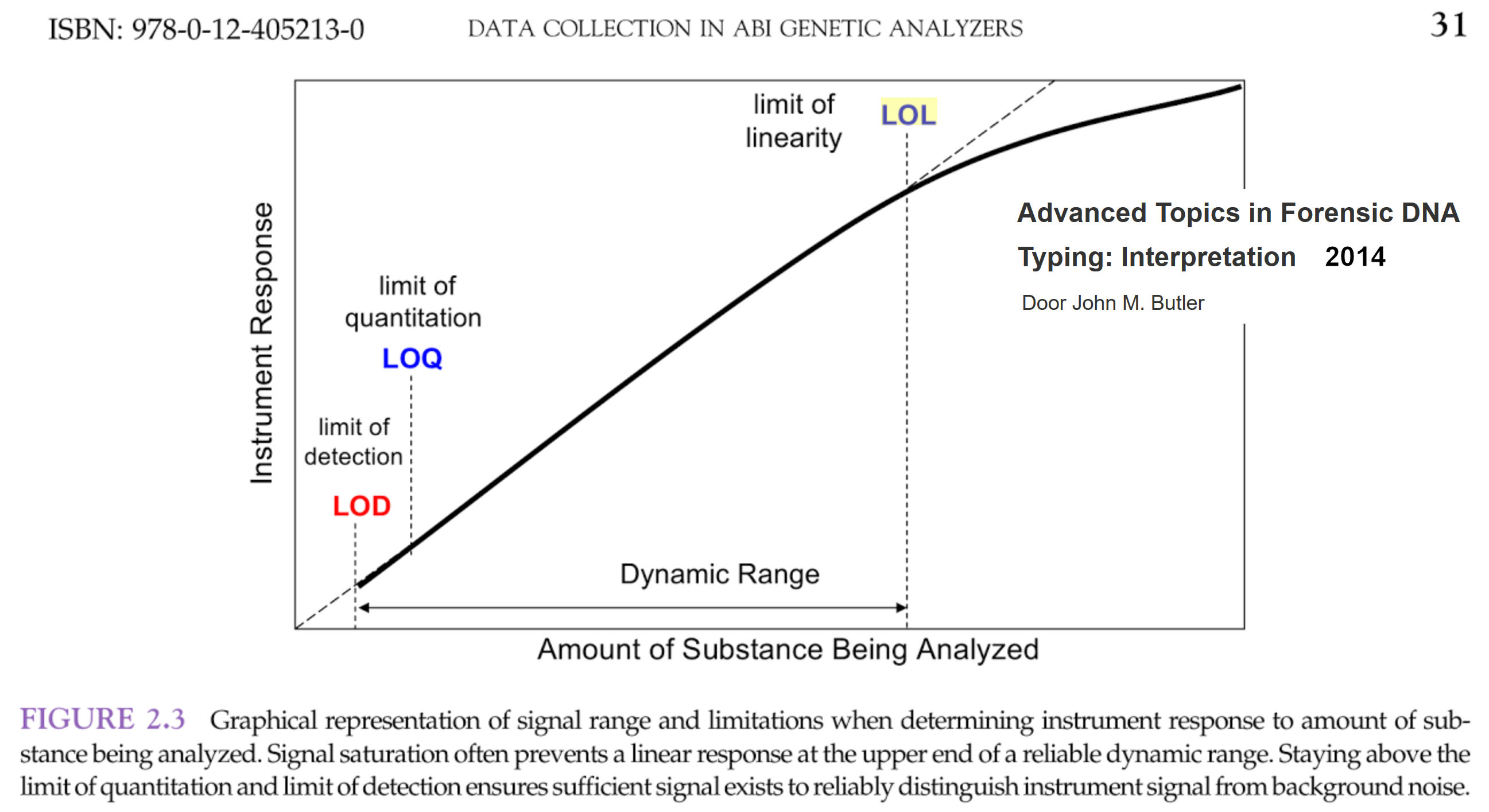 |
|
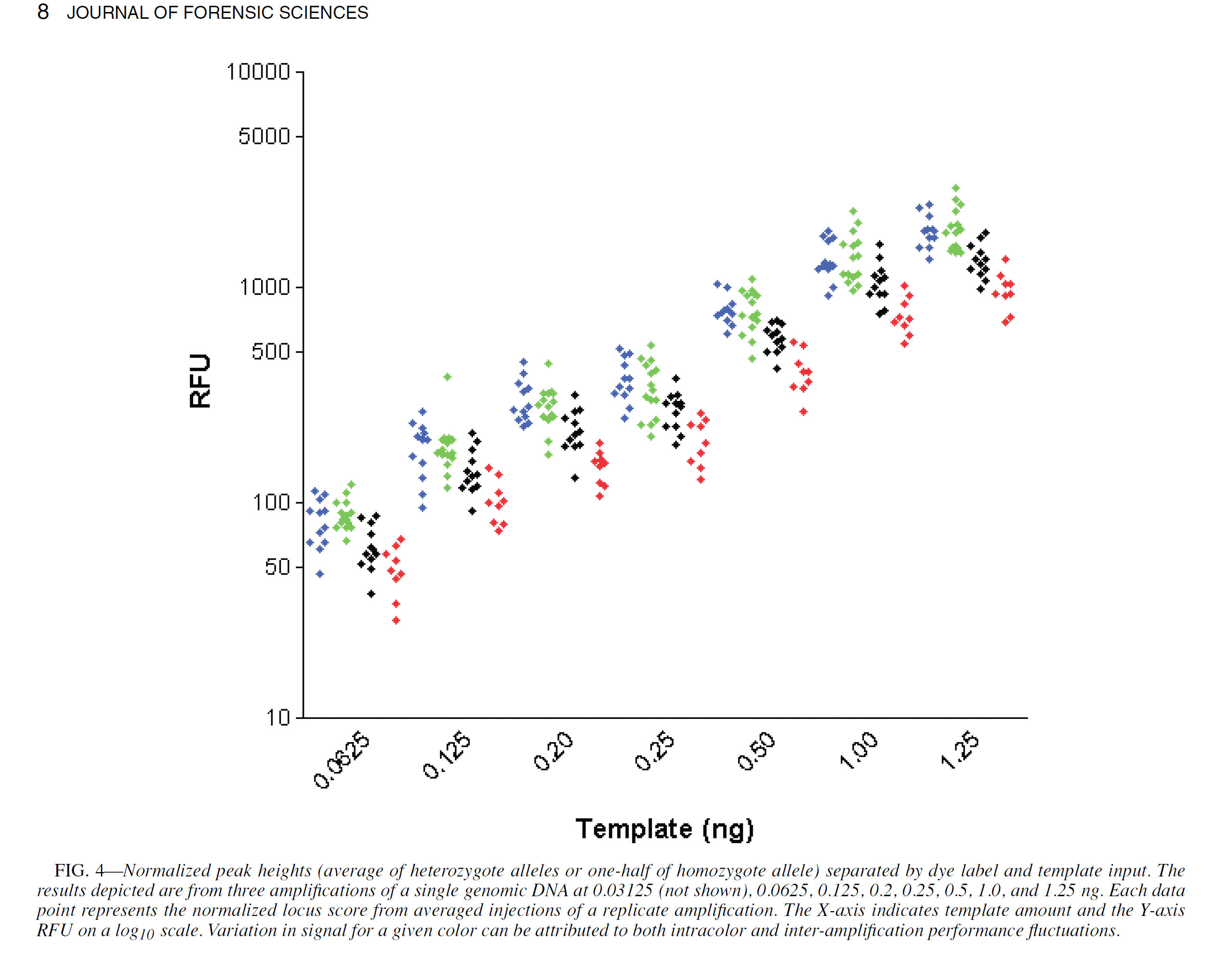
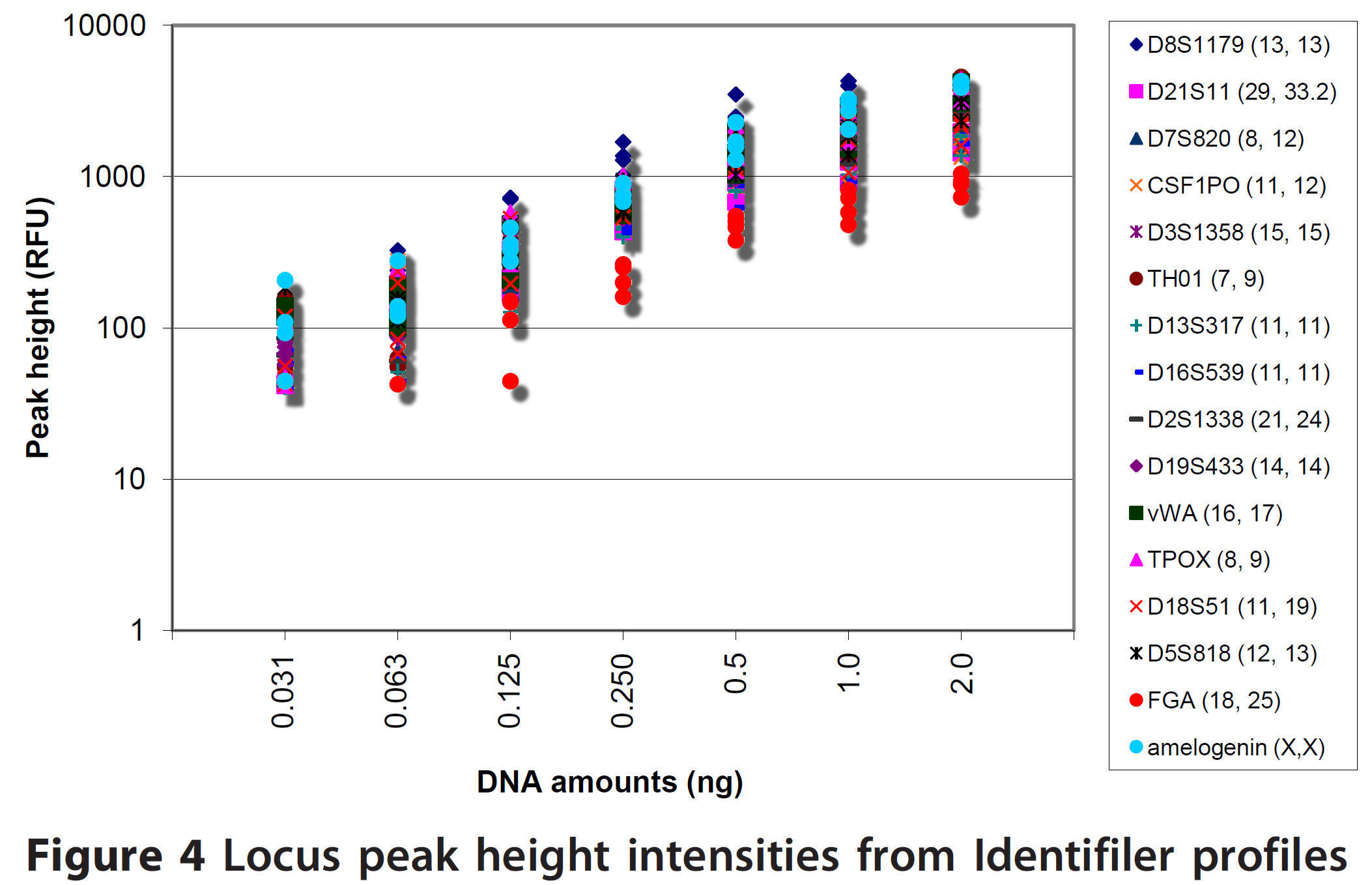
Twee
demonstraties
van de evenredigheid van de sterkte van profielen en de
hoeveelheid gemeten DNA. Links de uitleg van Butler, zo
ongeveer de geestelijke vader van de capillaire electroforese.
Rechts een afbeelding uit de manual van het door het NFI
gehanteerde systeem.
|
Zoals hierboven zichtbaar is, is er een
duidelijke relatie tussen de hoeveelheid DNA die in het PCR-apparaat wordt
gebracht en de uiteindelijke waarden van het DNA-profiel in het
electroferogram, zoals bij wijze van voorbeeld in paragraaf 6.3.1. is
getoond. Zoals daar is uiteengezet, vertoont zo'n electroferogram een
merkwaardige bijzonderheid, het diagram valt naar rechts toe gezien sterk
af in piekhoogten. Afgaand op de gebruikte techniek is dat absoluut niet
verrassend.
|
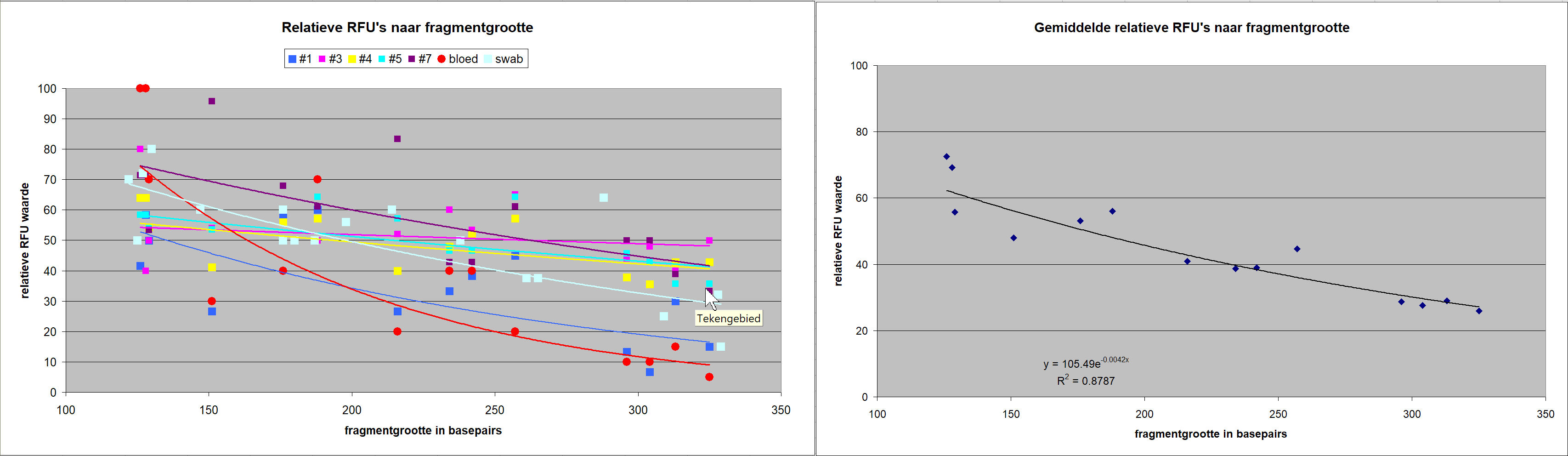
|
|
Analyse van de
RFU-waarden in resultaten van het NFI in de Deventer
Moordzaak in een aantal eenduidige sporen, vervaardigd met
dezelfde technieken.
|
In de voorgaande diagrammen is
gedemonstreerd, dat ook in de Deventer Moordzaak de electroferogrammen van
links naar rechts afvallen. Niet allemaal op dezelfde manier, maar wel
allemaal, waarbij opvalt dat het meest verse spoor - het kort
daarvoor afgenomen wangslijmvliesspoor van Louwes (swab) - net zo sterk
afneemt als het gemiddelde van de overige - ruim drie jaar oudere -
sporen.
Methode
Gebruikmakend van twee aspecten, de goede
correlatie binnen dezelfde locus en het aspect, dat de kleinste fragmenten
de duidelijkste representatie geven, heb ik per zogenaamde lane steeds ÚÚn
locus geselecteerd. Daarbij kwam de prettige omstandigheid om de hoek
kijken, dat de pieken van zowel het slachtoffer als de heer Louwes in de
heel duidelijke locus met de fraaie codering D3S1358 (blue lane)
homozygoot zijn, dus steeds naar verhouding tweemaal zo groot als
vergelijkbare andere pieken. Voorts is D8S1179 (green lane) gebruikt, die
alweer voor het slachtoffer homozygoot is (de heterozygote pieken van
Louwes heb ik opgeteld), evenals het locus D19S433 (yellow lane, dat
tweemaal heterozygoot is).
Als we alle resultaten voor de relevante sporen met elkaar vergelijken,
ontstaat er een zeer consistent plaatje:
|
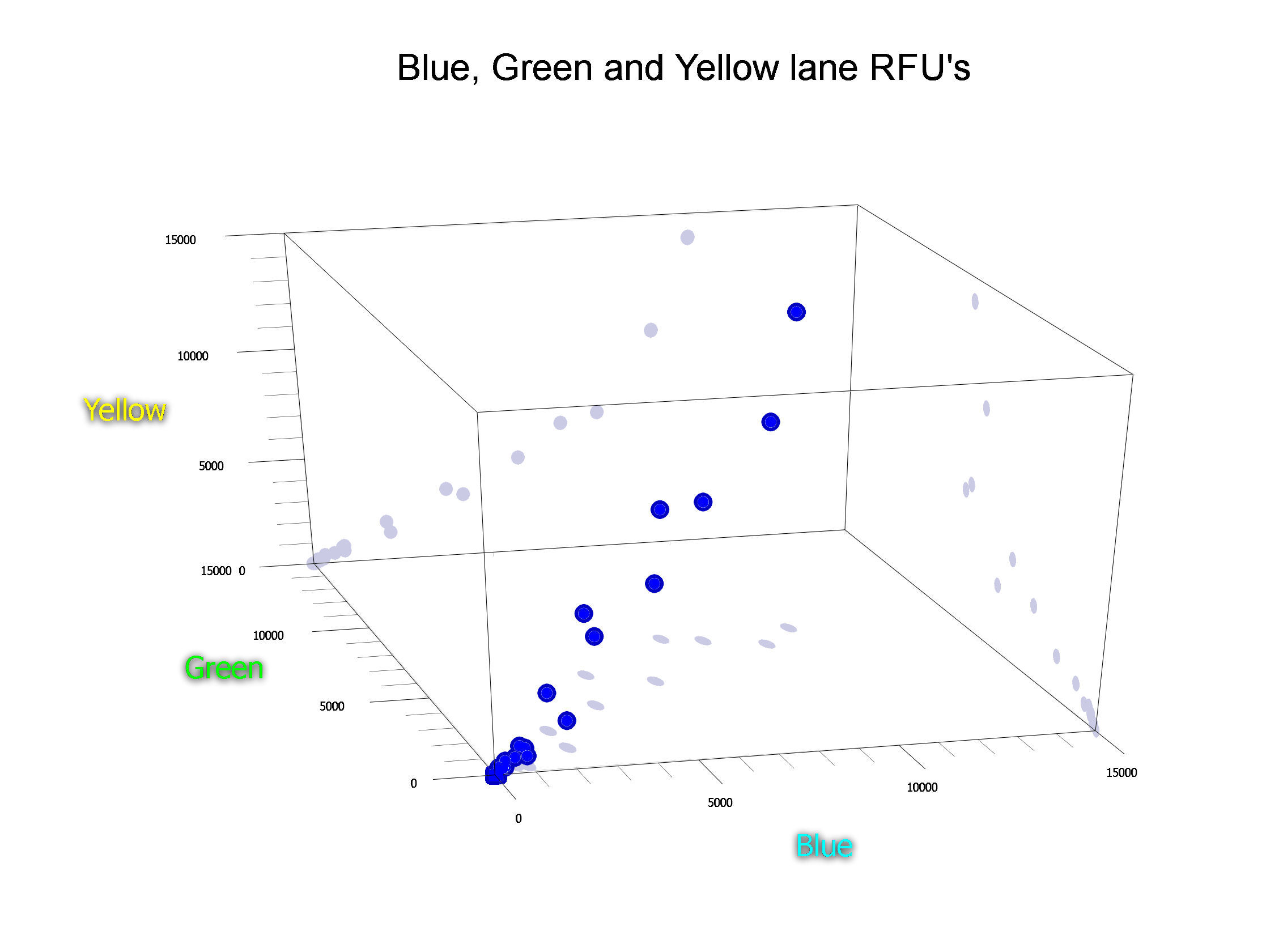
|
| De RFU-waarden van de drie
genoemde loci in de drie verschillende 'lanes' zijn hier - de
sporen in de Deventer Moordzaak - onderling gecorreleerd. |
Kortom, de overtuiging is ontstaan, dat
een goede inschatting kan worden gemaakt van de hoeveelheden DNA, die in
de verschillende sporen aanwezig waren, althans in onderlinge
vergelijking.
Er liggen nog steeds twee problemen op de weg, om geruimd te worden:
- In een enkel monster werd extra PCR-product ge´ntroduceerd
voorafgaande aan de electroforese
- en er werd vanaf spoor #11 een andere extractie-methode gebruikt.
Ik zal betogen dat 1. geen invloed op de analyses heeft, terwijl 2.
vrijwel onschadelijk is, omdat het DNA-onderzoek bijna geheel in twee
delen uiteen valt. Ten behoeve van de leesbaarheid breng ik deze
onderwerpen onder in een appendix.
In de volgende stap zijn de RFU-waarden gerelateerd aan de grootte van de
onderzochte sporen, zodat er in feite gekeken kan worden naar een
relatieve 2-dimensionale concentratie aan DNA in de verschillende sporen.
Hoe ervoor werd zorggedragen, dat de precieze oppervlakten werden bepaald,
behandel in ook in de appendix (photoshop- methode).
Resultaat
|
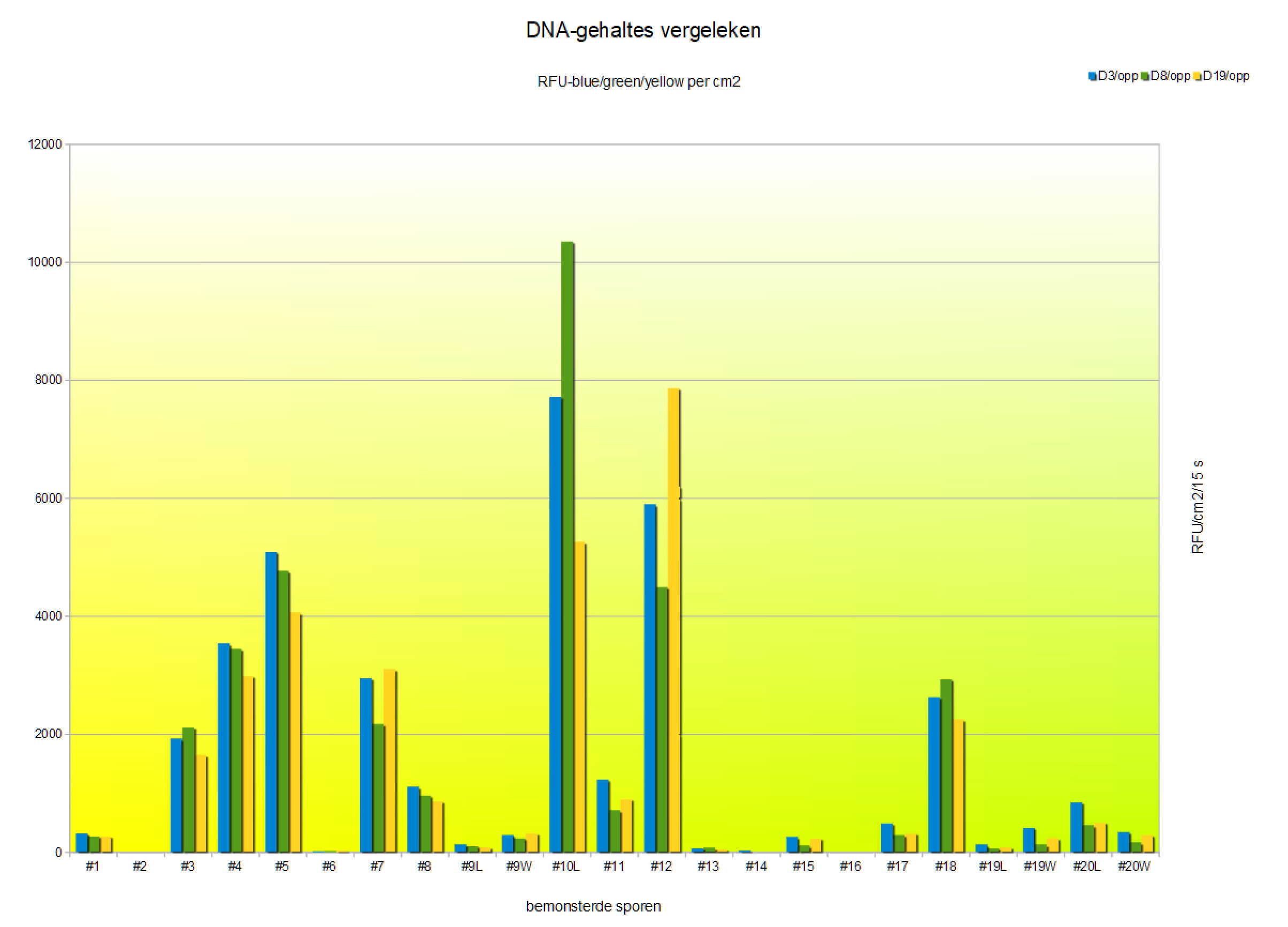
|
Voor ieder spoor zijn de
relatieve sterktes van de pieken in de drie eerder genoemde
loci uitgezet. In sommige gevallen zijn de bijdragen in een
spoor gesplitst naar donor of gespecificeerd naar donor (W
of L).
|
Wat in een eerste blik opvalt, is dat de
sporen waarover de meeste 'heisa' is ontstaan - de sporen #10 en #20 -
in het ene geval heel sterk uit de bus komt (#10) en in het andere geval
juist behoorlijk achterblijft (#20). In beide gevallen zal dit
aanleiding zijn de gebruikte hypotheses te verwerpen.
Dat gebeurt in de volgende paragrafen.
Appendices
De hoeveelheid ingebracht PCR-product.
Voor de capillaire electroforese wordt 1Ál
van het PCR-product in een monsterbuisje met water of formamide gebracht
en gebufferd, zodat de de zuurgroepen hun proton verliezen. Dat geldt voor
de door kopiŰren gevormde DNA-fragmenten, maar ook voor de ongebruikte
bouwstenen, die ook van nature vrijwel net zo zuur zijn. Brengt men meer
PCR-product in het monsterbuisje, dan vermeerdert men automatisch ook de
concurrentie. De concurrentie bestaat uit kleinere moleculen, waardoor er
geen voordeel ontstaat voor de bedoelde DNA-fragmenten, die meer dan 100x
zo omvangrijk zijn.
 |
|
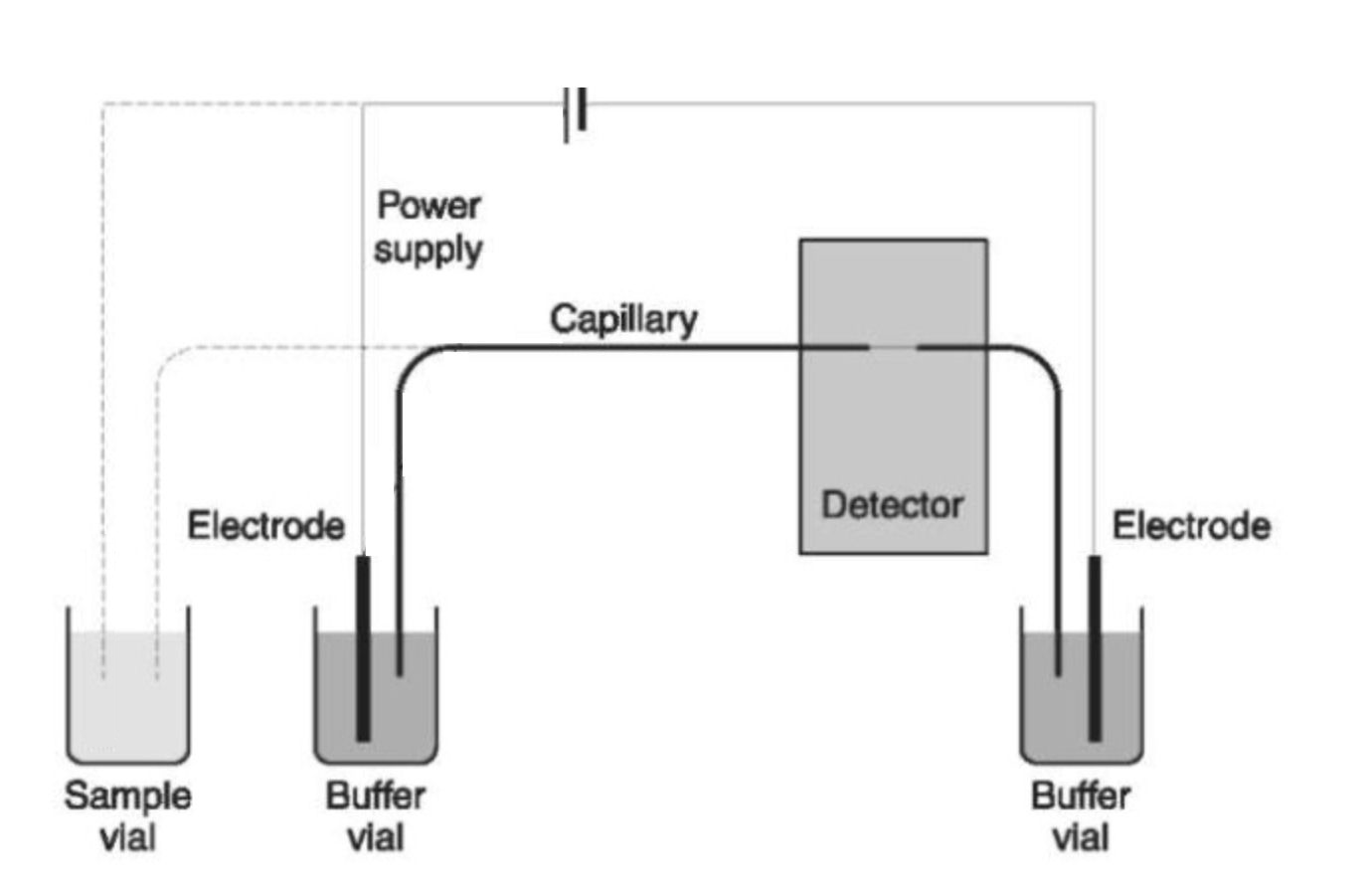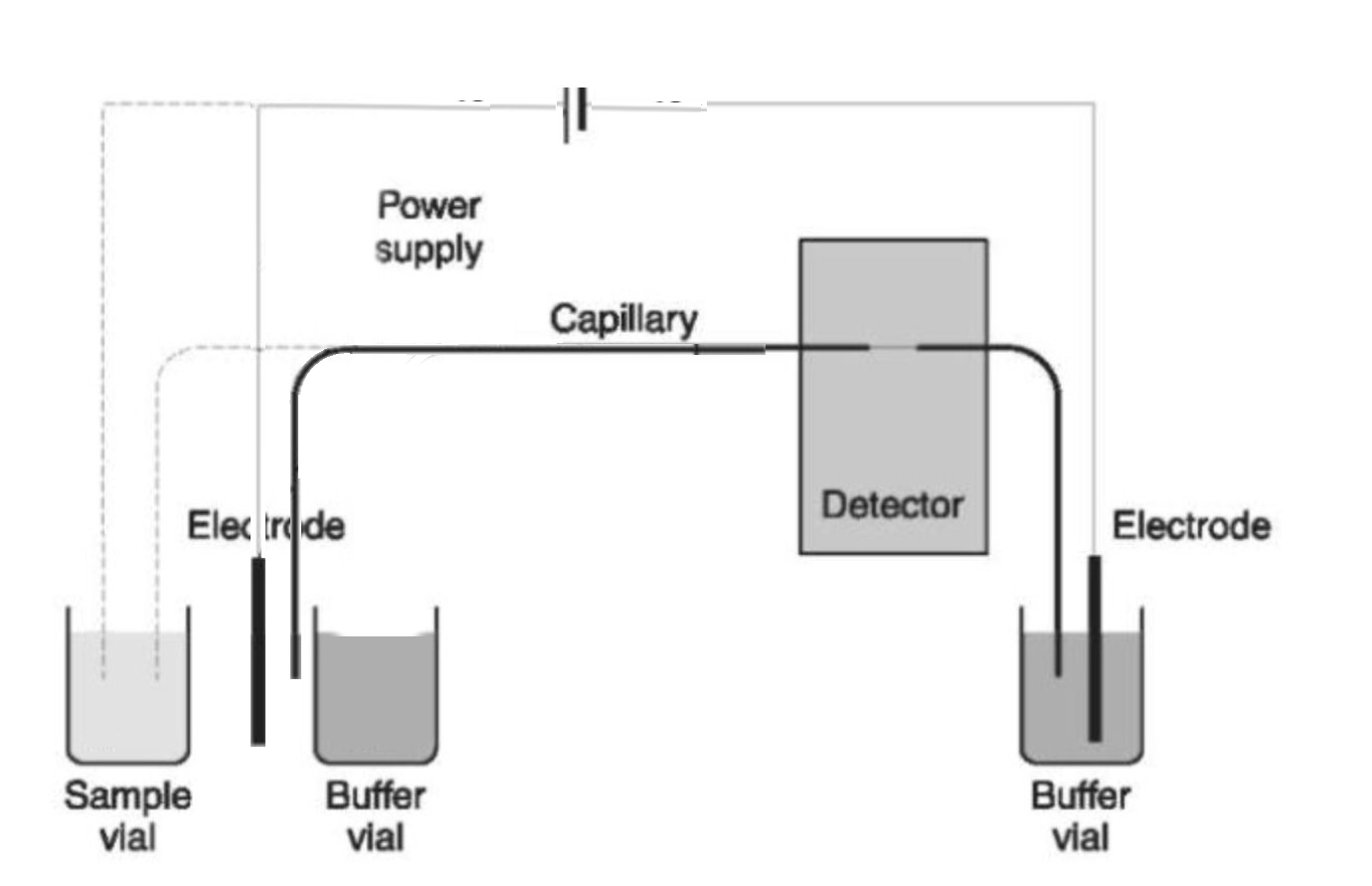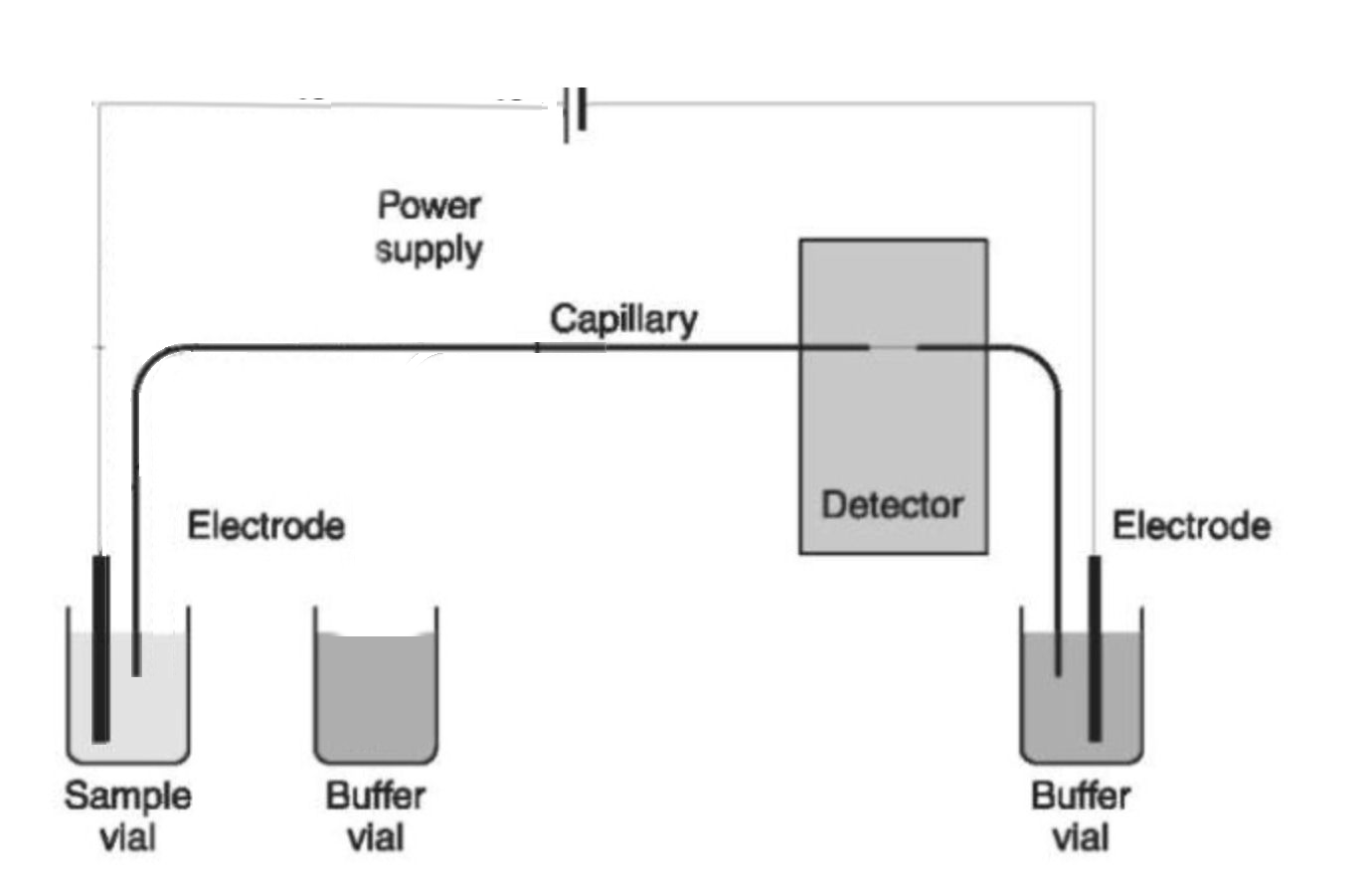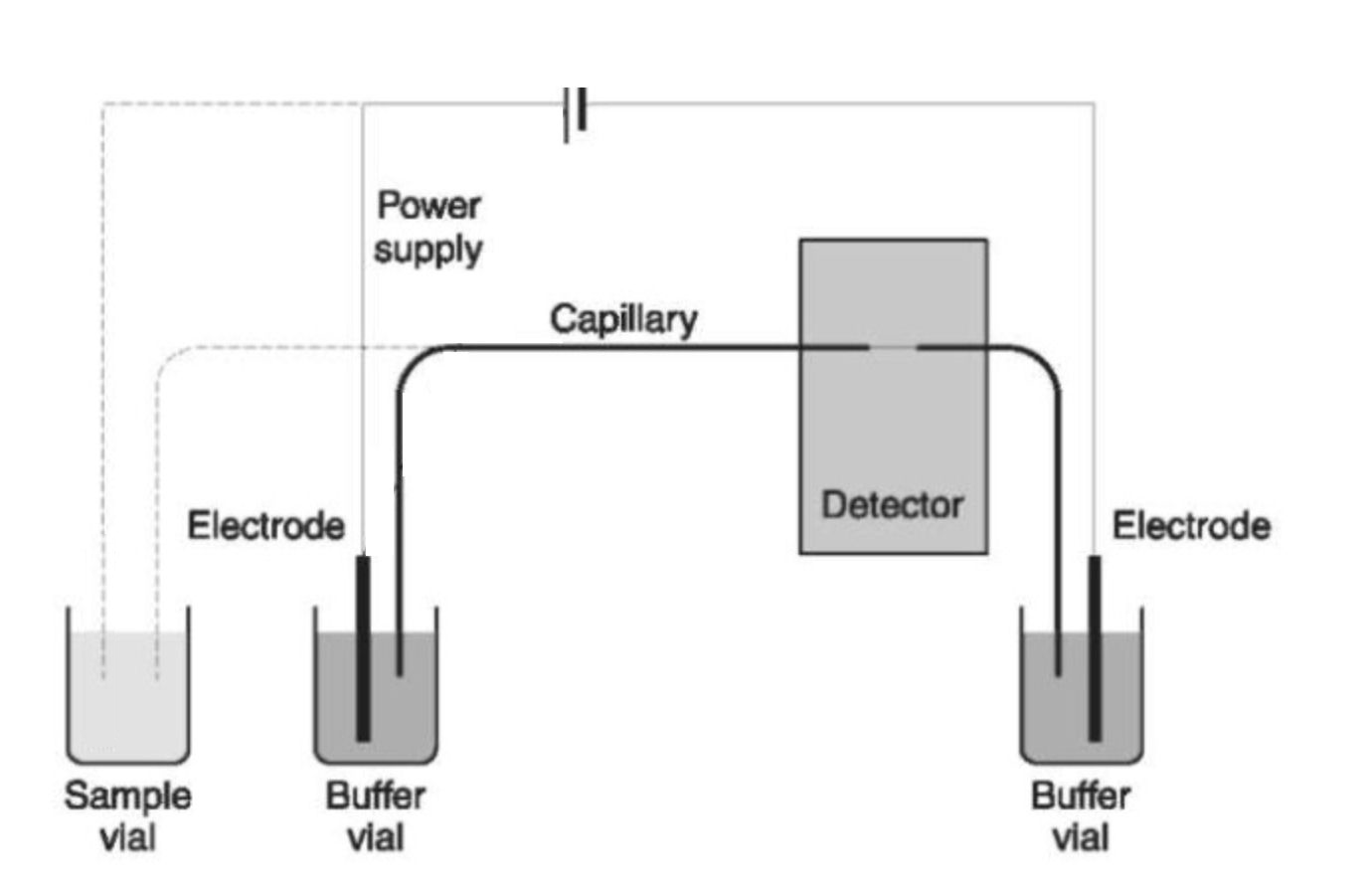
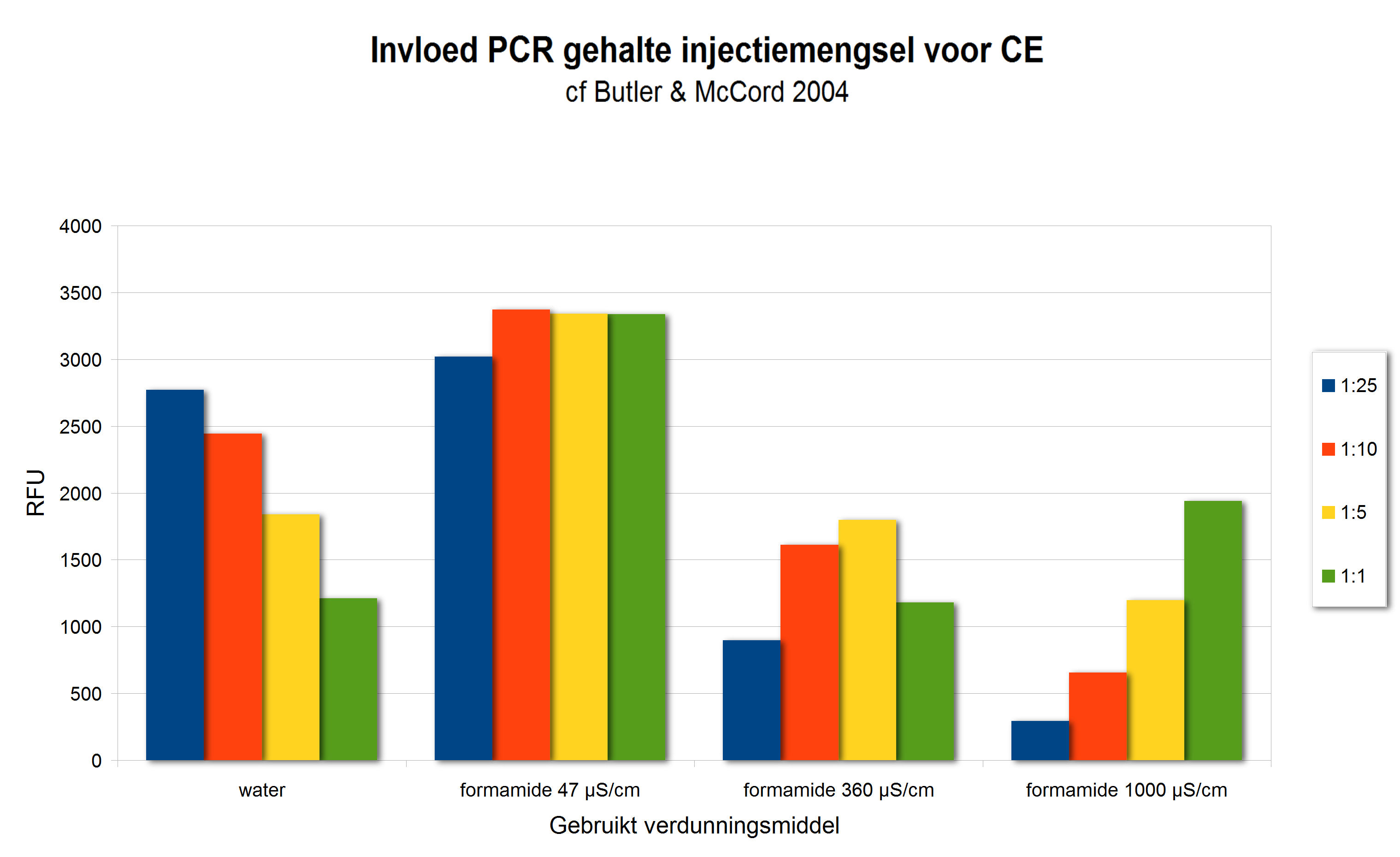
Links
de werking van de electrokinetische injectie. Tijdelijk worden
electrode en capillair in het monsterflesje gedompeld om wat
ionen uit het mengsel te vissen. Rechts een onderzoek van
Butler (diagram nagetekend). Hier is te zien, dat het
aanbieden van meer PCR-product contraproductief werkt, tenzij
men werkt met inferieure oplosmiddelen (rechter metingen).
|
De monstername aan de start van de
electroforese vindt plaats door het aanleggen van een spanningsveld tussen
(een electrode in) het monsterflesje en het uiteinde van de capillair,
waarin zich een scheidende gel bevindt. In feite vindt er op dat moment
'in het klein' een extra electroforese plaats: er wordt steeds een
overmaat aan meer kleine deeltjes in de gel opgenomen, ÚÚn van de oorzaken
voor het 'afvallen' van de diagrammen, zoals eerder is gedemonstreerd (zie
het voorbeeld in paragraaf 6.3.1.). De hoeveelheid opgenomen PCR-product
wordt bepaald door de heersende stroomsterkte en de duur van het
bemonsteren, de zogenaamde injectietijd (Q = i * t). De hoeveelheid ionen
die beschikbaar is, maakt in principe geen deel uit van deze formule. er
kunnen nooit te weinig ionen zijn, want dat is bijvoorbaat geregeld in de
samenstelling van het mengsel, dat in de PCR wordt samengebracht. Voor
eventuele verschillen in injectietijd t heb ik wel consequent
gecorrigeerd. De hoeveelheid PCR-product variŰren helpt dus niet, hetgeen
het NFI zelf heeft geverifieerd!
 |
|
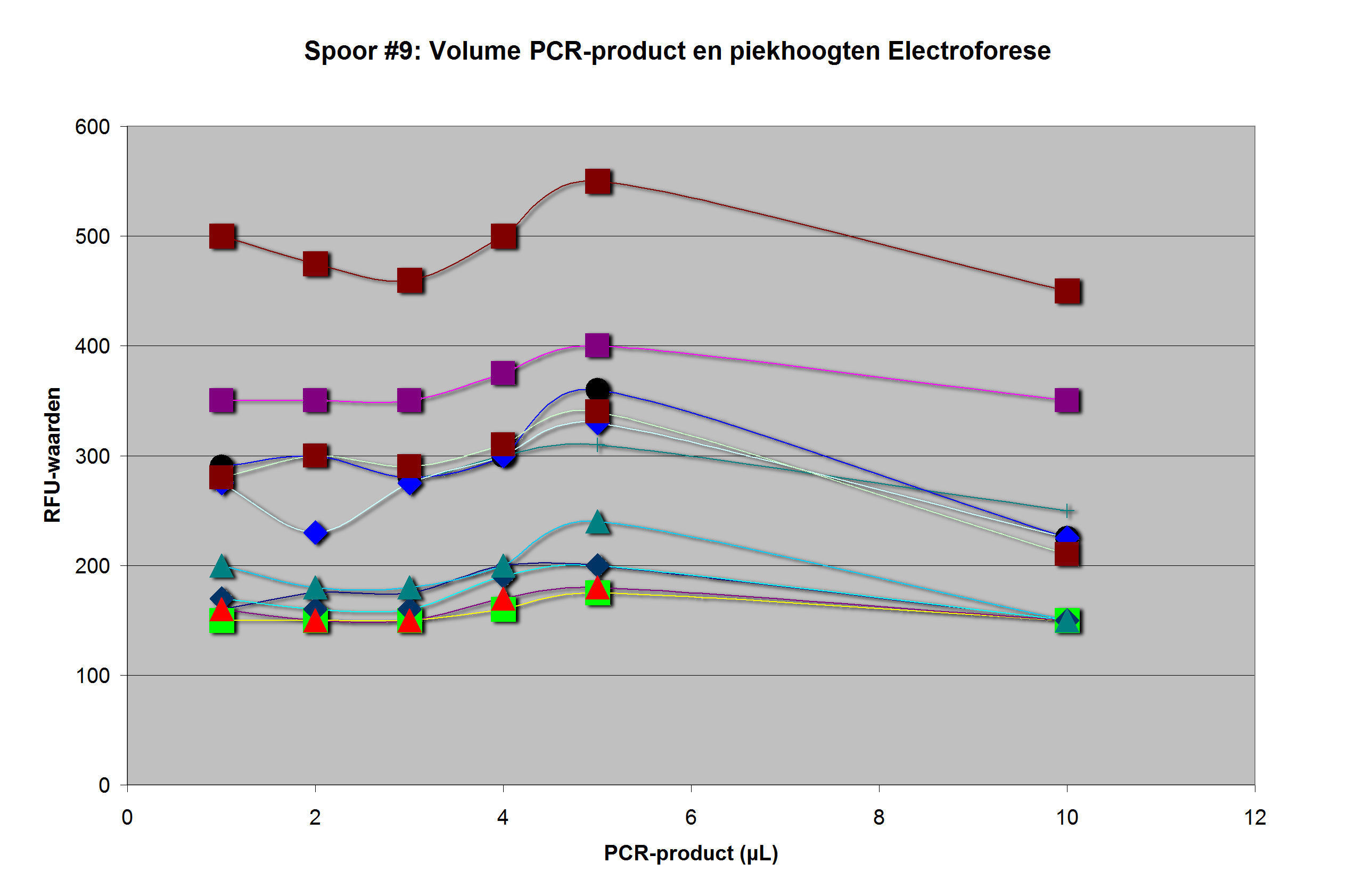
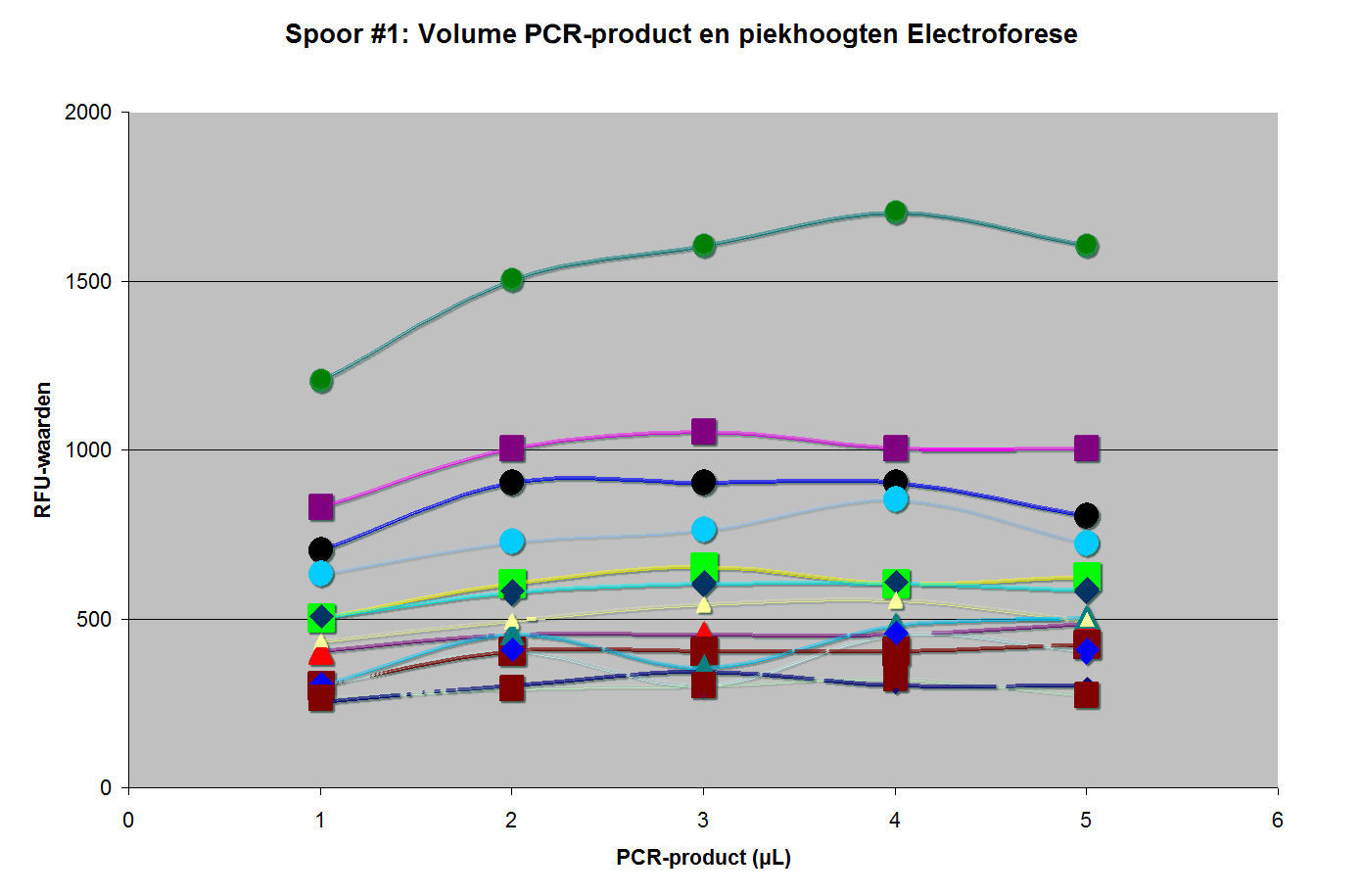
Twee
onderzoeken binnen een onderzoek. Hier onderzocht het NFI de
mogelijkheid een betere opbrengst aan DNA-pieken te krijgen
met grotere volumina aan PCR-product, ondanks dat al lang
bekend en aangetoond was (bijvoorbeeld door de eerder genoemde
Butler), dat dit niet werkt.
|
De gebruikte extractie-methodes
Er werden twee extractie-methoden gebruikt,
Chelex voor de eerste 10 sporen en QIAamp voor de overige 10. Het verschil
in resultaat tussen beide methoden laat zich als volgt karakteriseren:
- Chelex bevat meer DNA in een aanzienlijk groter volume (400 Ál)
- QIAamp bevat (iets) minder DNA bij een aanzienlijk grotere
concentratie (volume 50 - 100 Ál)
Omdat in beide gevallen ruimschoots
voldoende voldoende volume wordt geproduceerd voor het PCR-proces, is
QIAamp dus gewoon effectiever.Bij het vergelijken van sporen uit de reeks
1 -10 met de reeks 11 - 20 moet hier rekenschap van gegeven worden.
Photoshop
Met Adobe Photoshop Elements is het mogelijk
een aantal bepalingen te doen, die zeer behulpzaam zijn bij het bepalen
van een aantal kenmerken van de genomen monsters. De gelukkige
omstandigheid doet zich voor, dat vrijwel alle monsters zijn
gefotografeerd vˇˇr en na het uitknippen. Daarmee valt bijvoorbeeld vast
te stellen, hoe groot een bloedvlek was, en welk deel ervan in de
bemonstering kwam. Daarnaast bezit de blouse een bijzonder nuttig kenmerk:
er staat een stippeltjespatroon op met nauwkeurig bekende afmetingen. Door
steeds de virtuele oppervlakte van een zeshoek op de blouse te meten en te
vergelijken met de werkelijke oppervlakte van deze zeshoek (4.06 cm2
groot), kan iedere oppervlak worden geijkt, of de blouse nu recht van
boven is gefotografeerd of onder een hoek.
|

|
Verschillende stadia van
het Photoshop onderzoek. Zodra een gekleurd vak wordt
geselecteerd, kun je in Photoshop van die selectie het aantal
pixels en de luminantie van die pixels aflezen (beide met de
optie [Image] > [Histogram...]).
|
Zie toe, dat er voor spoor #10 in dit
voorbeeld twee manieren zijn om de oppervlakte van het spoor te
definiŰren.