NFI-hypothese spoor #20
In de bespreking van spoor #20 tijdens
het revisieproces van Den Bosch (2003-4) refereert ing. R. Eikelenboom
al dan niet bewust aan het verschijnsel geprogrammeerde celdood
(apoptosis), zoals dat (wellicht) optreedt bij de cornificatie van de
huid:
De kans op verkrijging van een
DNA-profiel speelt ook een rol. Uit bloed, sperma of speeksel wordt in
het algemeen vrij eenvoudig een DNA -profiel verkregen. Bij
aanrakingssporen of huidcellen is die kans erg klein. Weliswaar laat
de huid continu cellen los, maar deze bevatten weinig DNA -materiaal,
aangezien het om afstervende cellen gaat. Indien door de dader een
behoorlijke mate van kracht is uitgeoefend, is de kans dat er uit
huidcellen een DNA -profiel wordt verkregen, groter.
En:
Huidcellen die loskomen, zijn
afgestorven. Het DNA -materiaal in zo'n cel is grotendeels afgebroken.
Bij het gebruik van de Low Copy Number -methode zijn maar enkele
cellen nodig om een bruikbaar DNA -profiel te verkrijgen. Voor het met
behulp van de standaardmethoden, die door het NFI bij het DNA
-onderzoek worden gehanteerd, verkrijgen van een bruikbaar DNA-profiel
van huidcellen dienen minimaal 200 cellen te zijn
overgebracht. Bij zakelijk, oppervlakkig contact zoals het geven van
een hand of het voeren van een gesprek op geringe afstand tussen
personen wordt in het algemeen minder dan deze hoeveelheid
overgedragen.
Het Hof ging hierin mee, gelezen het
volgende:
Ing. Eikelenboom heeft in dit verband
in de eerste plaats onder meer opgemerkt dat de in het onderhavige
onderzoek verkregen DNA-profielen zijn bepaald met de
standaardmethoden die door het NFI bij het DNA-onderzoek worden
gehanteerd. Bij die methoden zullen over het algemeen geen profielen
worden verkregen uit celmateriaal dat kan worden overgedragen bij
zakelijk, oppervlakkig contact zoals het geven van een hand of het
voeren van een gesprek op geringe afstand tussen personen. Ter zitting
van 26 januari 2004 heeft ing. Eikelenboom verklaard dat voor het met
behulp van genoemde standaardmethoden verkrijgen van een bruikbaar
DNA-profiel van huidcellen minimaal 200 cellen dienen te zijn
overgebracht en dat bij het bedoelde zakelijke, oppervlakkige contact
in het algemeen minder dan deze hoeveelheid zal worden
overgedragen.(..) Ing. Eikelenboom heeft in dit verband in de eerste
plaats onder meer opgemerkt dat de in het onderhavige onderzoek
verkregen DNA-profielen zijn bepaald met de standaardmethoden die door
het NFI bij het DNA-onderzoek worden gehanteerd. Bij die methoden
zullen over het algemeen geen profielen worden verkregen uit
celmateriaal dat kan worden overgedragen bij zakelijk, oppervlakkig
contact zoals het geven van een hand of het voeren van een gesprek op
geringe afstand tussen personen. Ter zitting van 26 januari 2004 heeft
ing. Eikelenboom verklaard dat voor het met behulp van genoemde
standaardmethoden verkrijgen van een bruikbaar DNA-profiel van
huidcellen minimaal 200 cellen dienen te zijn overgebracht en
dat bij het bedoelde zakelijke, oppervlakkige contact in het algemeen
minder dan deze hoeveelheid zal worden overgedragen.
Mogelijkerwijs geeft ing. R. Eikelenboom in zijn bespreking van het
DNA-gehalte van huidcellen een verklaring voor de tienvoudige ophoging
van de minimaal benodigde hoeveelheid DNA (200 i.p.v 20 cellen),
waarvan hij melding maakte. Door de DNA-deskundige van het NFI (Dr. A
Kloosterman 2000) was vastgesteld dat de optimale DNA-input voor het
rond 2000 in gebruik genomen SGM-plus-systeem tussen de 30 en 150
cellen lag. Een stageverslag, gemaakt onder auspiciën van dezelfde
Dr. A. Kloosterman in 2003 bij het NFI meldde een ondergrens van 100
picogram/18 cellen (Emily Besselink, UvA/NFI 2003). Daar wordt
echter door geen van de betrokkenen - dus inclusief Dr. A. Kloosterman -
voor het Hof melding gemaakt. Het kan natuurlijk ook, dat ing.
Eikelenboom de begrippen cel en picogram verwarde; de detecteerbaarheid
van DNA via standaardprocedures werd destijds veelal rond de 100 tot 200
picogram gelegd (= rond de 20 cellen). Een door ing. Eikelenboom
aangedragen literatuurverwijzing meldde die getallen nadrukkelijk:
"Therefore, as few as 20 cells
will be sufficient to produce a DNA type profile. The skin surface
represents a large potential for a source of DNA profiles." (R. A.
Wickenheiser 2002).
Waarschijnlijk was deze zin door geen van
de deelnemers aan het proces gelezen. In ieder geval is de redenering
van ing. R. Eikelenboom ronduit speculatief. Dat valt uit meerdere
gezichtshoeken te benaderen:
- Stand van wetenschap op moment van de verklaring.
Alle artikelen omtrent van cornificatie (vorming huidcellen) in
relatie tot geprogrammeerde celdood melden een onvolledig begrip van
het mechanisme of mechanismen (cf. Eckhart et al. 2013). Dit geldt
zowel voor literatuur voor, als ten tijde van de besproken uitspraken.
Dit geldt trouwens ook nog nu, cf. Clare Rogerson et al. 2018.
- Cornificatie leidt tot het onzichtbaar worden van celkernen. Dat is
wat anders dan het verdwijnen van het DNA. DNA omvat 10% van de massa
van een celkern. Celkernen zijn –onder de microscoop- zichtbare
structuren, opgebouwd uit miljoenen moleculen. Individuele
DNA-moleculen zijn meestal vrijwel onzichtbaar en vertonen een
betrekkelijk hoge stabiliteit gezien het aantreffen van DNA in de
fossielen van bijvoorbeeld neanderthalers. Er zijn aanwijzingen dat
het DNA vrijkomt en daardoor juist gemakkelijker overdraagbaar wordt
(Kita et al. 2008). Volgens het mechanisme van apoptosis worden de
afbraakresten opgenomen door fagocyten en blijven dan gewoon
detecteerbaar (tijdens DNA-typering worden de fagocyten eenvoudig weer
afgebroken en komt het DNA vrij). Of apoptosis bij cornificatie een
rol speelt is intussen maar de vraag: Lippens et al. 2005. Fischer et
al. 2007 concluderen dat in ieder geval een soortgelijk proces
optreedt.
- Het verlies van celkernen bij cornificatie treedt niet bij iedereen
op. Indien betrokkene lijdt aan parakeratose (zoals bij roos en
psoriasis), blijven de celkernen typisch wél aanwezig (cf. Fischer et
al. 2017). In combinatie met het dan veelvuldig ontstaan van
huidschilfers (niet onrealistisch, Louwes is kaalhoofdig), wordt
DNA-overdracht alleen maar waarschijnlijker. Deze mogelijkheid werd
echter geheel buiten beschouwing gelaten.
- Afbraak van DNA wordt beschouwd als de onderdeel van geprogrammeerde
celdood. Deze afbraak vindt – naar de huidige stand van wetenschap –
niet willekeurig plaats, maar op bepaalde plaatsen in het DNA,
zogenaamde restriction sites. Veelal wordt dit toegeschreven aan het
enzym DNase1L2. Een gevolg hiervan is, dat bepaalde locaties van het
DNA niet gevoelig zijn voor deze vorm van afbraak. Daartoe behoren een
aantal sites, die gebruikt worden bij forensisch DNA-onderzoek
(Chun-nan Dong et. al 2016). Het is inderdaad zo, dat sommige van deze
locaties marginaal betere resultaten laten zien (zie de appendix),
maar dat is onvoldoende, om de 10-voudige afwijking in gevoeligheid,
die ing. Eikelenboom lijkt te hanteren, te verklaren. Het is in ieder
geval zo, dat bij deze DNA-afbraak brokstukken overblijven, die
typisch 200 of veelvouden van 200 baseparen lang zijn, dus in veel
gevallen gewoon geschikt zijn voor DNA-typering (die gebruik maakt van
fragmenten van 100 tot 340 baseparen lang).
Uiteindelijk alle problemen overziende,
lijkt maar één gevolgtrekking mogelijk: de uitspraken van ing. Eikelenboom
ontberen de benodigde wetenschappelijke onderbouwing. Het Hof had dit
kunnen weten op basis van het C.V. van ing. Eikelenboom, dat aangaf dat
hij op dit terrein slechts hts-diploma's bezat, dus volstrekt onvoldoende
basis om zijn analyses te rechtvaardigen. Een opvallend detail zal ik hier
niet ongenoemd laten: het desbetreffende rapport van ing. Eikelenboom werd
niet mede-ondertekend door de wel op dit terrein bevoegde Dr. Kloosterman.
appendix
Het DNA zit veilig opgeborgen in de celkern
doordat het periodiek om zogenaamde histonen opgewonden zit tot zgn.
nucleosomen (midden). Er ontstaat zo een soort kralenketting. De
kettingdraad tussen de kralen staan bekend als 'linker-DNA'. Dat
linker-DNA is het meest gevoelig voor enzymen, die het DNA kunnen klieven
(DNAse). Dan ontstaan stukken DNA met een typische lengte van 200
baseparen of veelvouden hiervan. Een aantal DNA-locaties, die gebruikt
worden bij forensisch DNA-onderzoek liggen in de afgeschermde gedeeltes,
andere liggen in het linker-DNA.
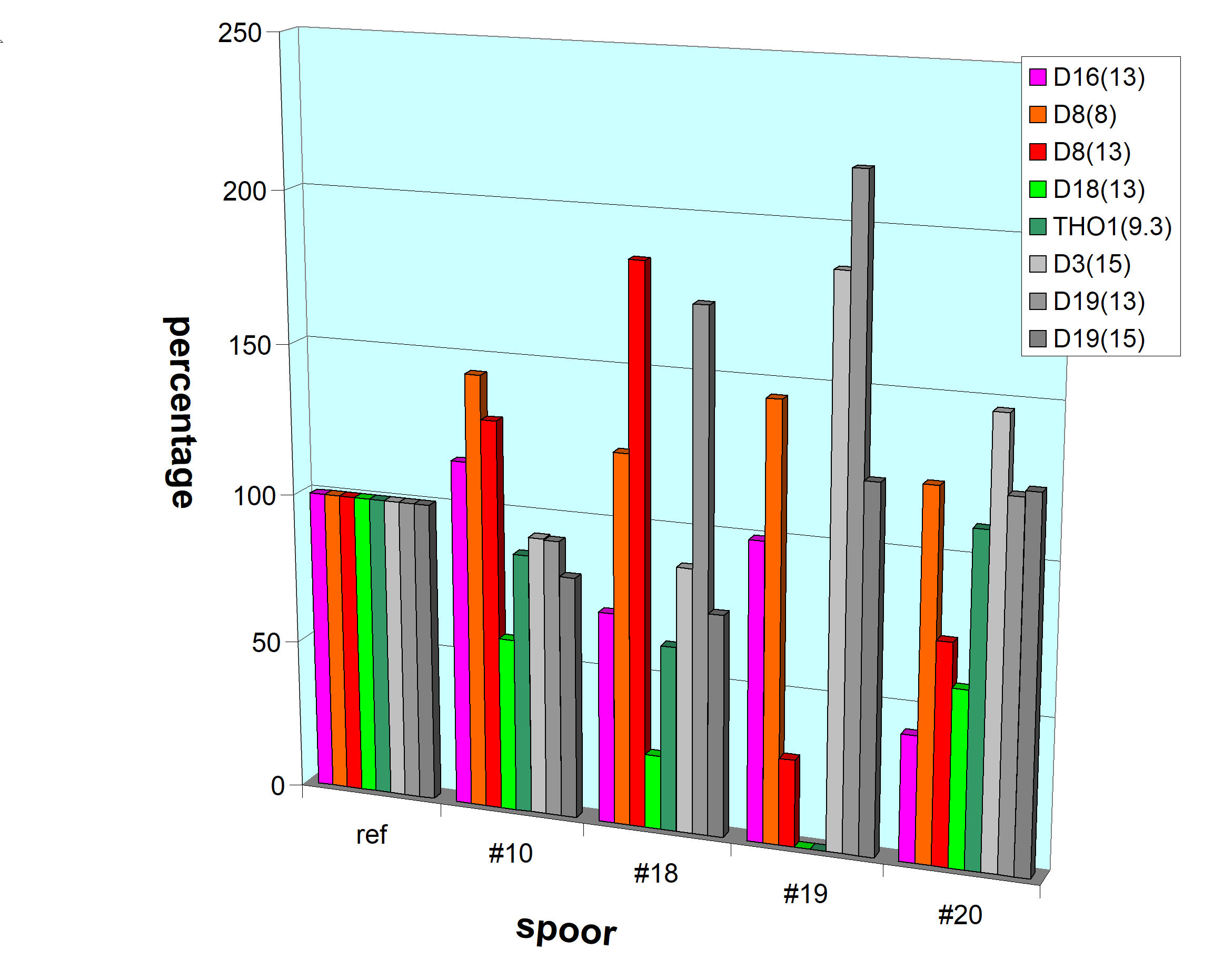 |
Van
vier hier relevante locaties is door Chun-nan Dong et al. 2016
vastgesteld of ze binnen een nucleosoom vallen (groen) of
binnen het 'linker-DNA' (rood). Hier zijn de signaalsterkten
van de pieken op deze locaties in diverse sporen van Louwes
vergeleken met de signaalsterkten in zijn referentiespoor (dat
dus 'intact' is). Ook zijn - in grijs - de responses van
locaties opgenomen, die gebruikt werden om de DNA-sporen in
sterkte met elkaar te vergelijken (zie 'naastliggende'
paragrafen). In spoor #20 is niet te zien, dat de 'beschermde'
locaties hogere responsies tonen, maar voor de verwante sporen
#18 en #19 geldt zelfs het tegendeel. Volgens de
huidcellen-hypothese zou het beeld hier duidelijk ten gunste
van de beschermde locaties (groen) moeten uitvallen. |
Gebruikte referenties:
Dr. A Kloosterman. DNA als gerechtelijk bewijsmateriaal. NFI 2000. http://bioplein.nl/attachments/File/Humane_Sporen/dna_a_g_b.pdf
E. Besselink. Current and Future Developments in Forensic DNA Typing.
Scriptie UVA/NFI. 2003. https://pdfs.semanticscholar.org/8f6f/df8c77d08afcc5630bc2872990e23c07f318.pdf
Leopold Eckhart et al. Cell death by cornification. Biochimica et
Biophysica Acta 1833. 2013. https://doi.org/10.1016/j.bbamcr.2013.06.010
Clare Rogerson et al. Uncovering mechanisms of nuclear degradation in
keratinocytes: A paradigm for nuclear degradation in other tissues.
NUCLEUS 9-1. 2018. https://doi.org/10.1080/19491034.2017.1412027
Kita et al. Morphological study of fragmented DNA on touched objects.
Forensic Science International: Genetics 3-1. 2008. https://www.sciencedirect.com/science/article/abs/pii/S1872497308001336
Chun-nan Dong et al. Whole genome nucleosome sequencing identifies novel
types of forensic markers in degraded DNA samples. Nature Scientific
Reports 26101. 2016. http://dx.doi.org/10.1038/srep26101
Lippens et al. Death penalty for keratinocytes: apoptosis versus
cornification. Cell Death and Differentiation 12. 2005. https://www.nature.com/articles/4401722
Fischer et al. DNase1L2 Degrades Nuclear DNA during Corneocyte Formation.
Journal of Investigative Dermatology 127. 2007. https://doi.org/10.1038/sj.jid.5700503
Fischer et al. Inactivation of DNase1L2 and DNase2 in keratinocytes
suppresses DNA degradation during epidermal cornification and results in
constitutive parakeratosis. Nature Scientific Reports 7. 2017. https://www.nature.com/articles/s41598-017-06652-8
R. A. Wickenheiser. Trace DNA: A Review, Discussion of Theory, and
Application of the Transfer of Trace Quantities of DNA Through Skin
Contact. J Forensic Sci 47(3). 2002. https://www.pubfacts.com/detail/12051321/
